近日,北京大学分子医学研究所李川昀课题组与张秀琴研究员、中国科学院动物所张勇研究员等合作,在国际上首次获得恒河猴全编辑组(RNA Editome),包含三万多个RNA编辑位点,涵盖恒河猴多个体、多组织和多发育时期。进一步,他们通过恒河猴作为人类近缘物种的独特视角,从分子演化角度证明了“RNA编辑”这一基因调控新层次的整体功能性,并首次阐释了人类特有RNA编辑事件对人类转录组的调控。题为“RNA Editome in Rhesus Macaque Shaped by Purifying Selection”的研究论文在PLoS Genetics杂志发表,题为“Evolutionary Interrogation of Human Biology in Well-annotated Genomic Framework of Rhesus Macaque”的研究论文作为封面文章(Cover Story)在Molecular Biology and Evolution杂志发表。
RNA编辑可引起基因组与其编码的转录组在特定位点的序列差异,在个体发育、复杂疾病调控中发挥重要作用。然而,如何准确鉴定RNA编辑位点是该领域面临的一个技术挑战。已有多篇评论表明,2011年发表在Science杂志的人类编辑组集合中,90%以上是由技术误差导致的假阳性。在此背景下,人们只能把不同研究工作呈现出的编辑组异质性,笼统地归因于调控“复杂性”。李川昀课题组运用恒河猴作为模式动物和人类近缘物种的双重优势,结合深度测序、质谱和低通量验证,解决了RNA编辑鉴定中的多个技术难点,使恒河猴RNA编辑组实验验证率达到97%,呈现出高度的准确性、完整性和定量精度。同时,运用多组织、多个体数据,清晰地阐述了RNA编辑其实在很大程度上受控于ADAR基因介导的时空调控,澄清了该领域当前对RNA编辑调控“复杂性”的含糊不当的认识。
RNA编辑研究的另一个难点是其整体功能性。目前虽有个案研究揭示RNA编辑可以通过改变蛋白质的氨基酸组成从而在复杂疾病中发挥作用,但由高通量测序得到的数以万计的编辑事件是否在整体上具有生物功能,仍有待诠释。通过跨物种的比较基因组学研究,本项工作发现自然选择在维持RNA编辑组过程中发挥重要作用,为阐明RNA编辑的整体功能性提供了更具说服力的分子演化证据。研究同时明确了人类特有RNA编辑事件对人类转录组的调控,为揭示人类特有性状的分子基础提供了新视角。
上述两项进展得益于课题组前期研究的积累。他们前期建立的恒河猴基因组知识库RhesusBase,纠正了在1/3恒河猴基因结构注释中存在的错误,为在恒河猴中开展功能基因组学研究铺平了道路(Zhang et al, Nucleic Acids Res, 2013)。此外,课题组前期运用恒河猴模型开展的分子演化研究(Xie et al, PLoS Genet, 2012),从理论和方法学角度为研究RNA编辑的整体功能性奠定了必要的基础。
北京大学分子医学研究所三年级硕博连读研究生张仕坚、刘楚珺同学和于鹏博士为Molecular Biology and Evolution论文共同第一作者,四年级研究生陈加余同学为PLoS Genetics论文第一作者。本项研究受到国家973项目和国家自然科学基金等项目支持。
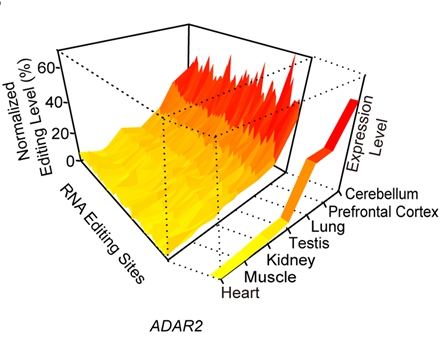
恒河猴全编辑组(PLoS Genetics, 2014; Mol Biol Evol, 2014)